Public Computational Chemistry Database(PCCDB)は、以前紹介しいた、PubChemQCの上位プロジェクトです。
PubChemQCの検索をさらに便利にできたり、色々なツールが利用できたりします。利用登録は必要ありません。無料で利用できるので是非活用してみましょう。

様々な分子情報を元に、PubChemQCに登録された分子を検索することができます。ちなみにPubChemQCは、量子化学計算で得られた構造最適化データに関するデータベースです(詳しくはこっちらの記事を参照)。
HOMO-LUMOギャップや各フロンティア軌道のエネルギー、励起エネルギー、LogPや融点などから検索できるので、ピンポイントで化合物をヒットさせることもできますし、材料や触媒設計・開発をする際に、候補化合物を見つけるのにも役立ちそうです。
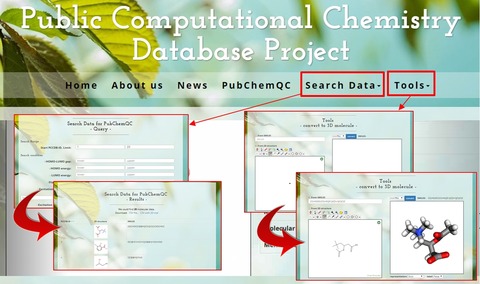
その他にも、トップページ上部の「Toolsボタン」からは色々と便利なツールを利用できます。分子の3Dビュアーを利用して、SMILES記法や2D構造から3D構造を得ることができます。3Dに変換した分子座標ファイルはダウンロードできるので、GAMESSなどの入力ファイル(.inp)の作成に利用できます。
2D構造から3D構造の分子座標を得たい場合は、以前紹介したMolViewの代替として使用するのも良いでしょう。また、検索結果をCSVファイルでダウンロードしたり、CSVファイルから検索することも可能です。
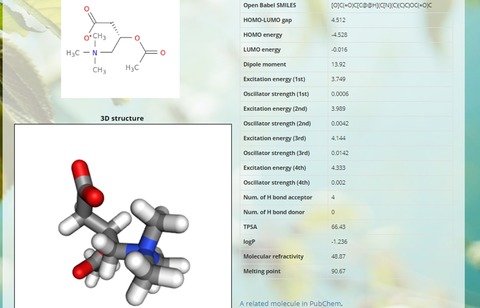
アセチル-L-カルニチンの分子情報と共に分子座標やMulliken(Lowdin)電荷、UVスペクトルなどの情報が表示されます。計算に使用したGAMESSのアウトプットファイル(最適化構造、励起状態)などは、ページの一番最後にダウンロードリンクがあります。
今後も、量子化学計算に関するデータベースの紹介を行っていきます。
【関連記事】
データベースを利用しよう その①~PubChemQC~
紫外可視吸収スペクトルを計算してみよう
構造最適化について知ろう
PubChemQCの検索をさらに便利にできたり、色々なツールが利用できたりします。利用登録は必要ありません。無料で利用できるので是非活用してみましょう。

どんなデータが利用できるの?
PCCDB(http://pccdb.org)様々な分子情報を元に、PubChemQCに登録された分子を検索することができます。ちなみにPubChemQCは、量子化学計算で得られた構造最適化データに関するデータベースです(詳しくはこっちらの記事を参照)。
HOMO-LUMOギャップや各フロンティア軌道のエネルギー、励起エネルギー、LogPや融点などから検索できるので、ピンポイントで化合物をヒットさせることもできますし、材料や触媒設計・開発をする際に、候補化合物を見つけるのにも役立ちそうです。

その他にも、トップページ上部の「Toolsボタン」からは色々と便利なツールを利用できます。分子の3Dビュアーを利用して、SMILES記法や2D構造から3D構造を得ることができます。3Dに変換した分子座標ファイルはダウンロードできるので、GAMESSなどの入力ファイル(.inp)の作成に利用できます。
2D構造から3D構造の分子座標を得たい場合は、以前紹介したMolViewの代替として使用するのも良いでしょう。また、検索結果をCSVファイルでダウンロードしたり、CSVファイルから検索することも可能です。
利用方法は?
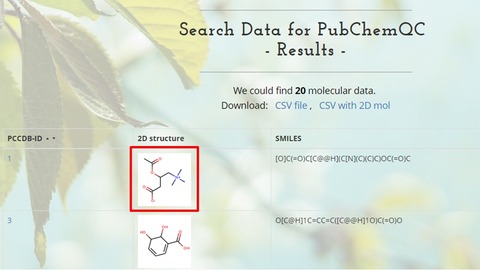
トップページのSearch Data >Queryを選択するとPubChemQCを詳細検索する画面が表示されます。試しに、一番下の融点を150~160℃で検索してみましょう。ヒットした候補化合物が表示されましたね。一番上のアセチル-L-カルニチンをクリックしてみましょう。
アセチル-L-カルニチンの分子情報と共に分子座標やMulliken(Lowdin)電荷、UVスペクトルなどの情報が表示されます。計算に使用したGAMESSのアウトプットファイル(最適化構造、励起状態)などは、ページの一番最後にダウンロードリンクがあります。

おわりに
今回は、PubChemQCの検索をより便利にするPublic Computational Chemistry Database(PCCDB)について紹介しました。2017年7/18に立ち上げられた新しいサイトなので、今後ますます使いやすくなっていくことが期待されます。今後も、量子化学計算に関するデータベースの紹介を行っていきます。
【関連記事】
データベースを利用しよう その①~PubChemQC~
紫外可視吸収スペクトルを計算してみよう
構造最適化について知ろう

コメント